Это небольшая аналитика, чтобы получить некоторое представление о хаосе, вызванном коронавирусом. Немного графики и статистики для общего представления.
Данные — Novel Corona Virus 2019 Dataset
Импортируем необходимые библиотеки.
import numpy as np # линейная алгебра
import pandas as pd # обработка данных, CSV afqk I/O (например pd.read_csv)
import matplotlib.pyplot as plt
import seaborn as sns
sns.set_context('paper')
import random
def random_colours(number_of_colors):
'''
Простая функция для генерации случайных цветов.
Входные данные:
number_of_colors - целочисленное значение, указывающее
количество цветов, которые будут сгенерированы.
Выход:
Цвет в следующем формате: ['#E86DA4'].
'''
colors = []
for i in range(number_of_colors):
colors.append("#"+''.join([random.choice('0123456789ABCDEF') for j in range(6)]))
return colors
Статистический анализ
data = pd.read_csv('/novel-corona-virus-2019-dataset/2019_nCoV_data.csv')
data.head()
| Sno | Province/State | Country | Last Update | Confirmed | Deaths | Recovered | |
|---|---|---|---|---|---|---|---|
| 0 | 1 | Anhui | China | 1/22/2020 12:00 | 1.0 | 0.0 | 0.0 |
| 1 | 2 | Beijing | China | 1/22/2020 12:00 | 14.0 | 0.0 | 0.0 |
| 2 | 3 | Chongqing | China | 1/22/2020 12:00 | 6.0 | 0.0 | 0.0 |
| 3 | 4 | Fujian | China | 1/22/2020 12:00 | 1.0 | 0.0 | 0.0 |
| 4 | 5 | Gansu | China | 1/22/2020 12:00 | 0.0 | 0.0 | 0.0 |
data.info()
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 497 entries, 0 to 496
Data columns (total 7 columns):
Sno 497 non-null int64
Province/State 393 non-null object
Country 497 non-null object
Last Update 497 non-null object
Confirmed 497 non-null float64
Deaths 497 non-null float64
Recovered 497 non-null float64
dtypes: float64(3), int64(1), object(3)
memory usage: 27.3+ KB
Растет метрик по числовым колонкам.
data.describe()
| Sno | Confirmed | Deaths | Recovered | |
|---|---|---|---|---|
| count | 434.000000 | 434.000000 | 434.000000 | 434.000000 |
| mean | 217.500000 | 80.762673 | 1.847926 | 1.525346 |
| std | 125.429263 | 424.706068 | 15.302792 | 9.038054 |
| min | 1.000000 | 0.000000 | 0.000000 | 0.000000 |
| 25% | 109.250000 | 2.000000 | 0.000000 | 0.000000 |
| 50% | 217.500000 | 7.000000 | 0.000000 | 0.000000 |
| 75% | 325.750000 | 36.000000 | 0.000000 | 0.000000 |
| max | 434.000000 | 5806.000000 | 204.000000 | 116.000000 |
Растет метрик по не числовым колонкам.
data.describe(include="O")
| Province/State | Country | Last Update | |
|---|---|---|---|
| count | 393 | 497 | 497 |
| unique | 45 | 31 | 13 |
| top | Ningxia | Mainland China | 1/31/2020 19:00 |
| freq | 10 | 274 | 63 |
Преобразуем данные Last Update в datetime
data['Last Update'] = pd.to_datetime(data['Last Update'])
Добавляем колонки Day и Hour
data['Day'] = data['Last Update'].apply(lambda x:x.day)
data['Hour'] = data['Last Update'].apply(lambda x:x.hour)
Данные только за 30 день января.
data[data['Day'] == 30]
| Sno | Province/State | Country | Last Update | Confirmed | Deaths | Recovered | Day | Hour | |
|---|---|---|---|---|---|---|---|---|---|
| 375 | 376 | Hubei | Mainland China | 2020-01-30 21:30:00 | 5806.0 | 204.0 | 116.0 | 30 | 21 |
| 376 | 377 | Zhejiang | Mainland China | 2020-01-30 21:30:00 | 537.0 | 0.0 | 9.0 | 30 | 21 |
| 377 | 378 | Guangdong | Mainland China | 2020-01-30 21:30:00 | 393.0 | 0.0 | 11.0 | 30 | 21 |
| 378 | 379 | Henan | Mainland China | 2020-01-30 21:30:00 | 352.0 | 2.0 | 3.0 | 30 | 21 |
| 379 | 380 | Hunan | Mainland China | 2020-01-30 21:30:00 | 332.0 | 0.0 | 2.0 | 30 | 21 |
| 380 | 381 | Jiangxi | Mainland China | 2020-01-30 21:30:00 | 240.0 | 0.0 | 7.0 | 30 | 21 |
| 381 | 382 | Anhui | Mainland China | 2020-01-30 21:30:00 | 237.0 | 0.0 | 3.0 | 30 | 21 |
| 382 | 383 | Chongqing | Mainland China | 2020-01-30 21:30:00 | 206.0 | 0.0 | 1.0 | 30 | 21 |
| 383 | 384 | Shandong | Mainland China | 2020-01-30 21:30:00 | 178.0 | 0.0 | 2.0 | 30 | 21 |
| 384 | 385 | Sichuan | Mainland China | 2020-01-30 21:30:00 | 177.0 | 1.0 | 1.0 | 30 | 21 |
| 385 | 386 | Jiangsu | Mainland China | 2020-01-30 21:30:00 | 168.0 | 0.0 | 2.0 | 30 | 21 |
| 386 | 387 | Shanghai | Mainland China | 2020-01-30 21:30:00 | 128.0 | 1.0 | 9.0 | 30 | 21 |
| 387 | 388 | Beijing | Mainland China | 2020-01-30 21:30:00 | 121.0 | 1.0 | 5.0 | 30 | 21 |
| 388 | 389 | Fujian | Mainland China | 2020-01-30 21:30:00 | 101.0 | 0.0 | 0.0 | 30 | 21 |
| 389 | 390 | Guangxi | Mainland China | 2020-01-30 21:30:00 | 87.0 | 0.0 | 2.0 | 30 | 21 |
| 390 | 391 | Hebei | Mainland China | 2020-01-30 21:30:00 | 82.0 | 1.0 | 0.0 | 30 | 21 |
| 391 | 392 | Yunnan | Mainland China | 2020-01-30 21:30:00 | 76.0 | 0.0 | 0.0 | 30 | 21 |
| 392 | 393 | Shaanxi | Mainland China | 2020-01-30 21:30:00 | 87.0 | 0.0 | 0.0 | 30 | 21 |
| 393 | 394 | Heilongjiang | Mainland China | 2020-01-30 21:30:00 | 59.0 | 2.0 | 0.0 | 30 | 21 |
| 394 | 395 | Hainan | Mainland China | 2020-01-30 21:30:00 | 50.0 | 1.0 | 1.0 | 30 | 21 |
| 395 | 396 | Liaoning | Mainland China | 2020-01-30 21:30:00 | 45.0 | 0.0 | 1.0 | 30 | 21 |
| 396 | 397 | Shanxi | Mainland China | 2020-01-30 21:30:00 | 39.0 | 0.0 | 1.0 | 30 | 21 |
| 397 | 398 | Tianjin | Mainland China | 2020-01-30 21:30:00 | 32.0 | 0.0 | 0.0 | 30 | 21 |
| 398 | 399 | Gansu | Mainland China | 2020-01-30 21:30:00 | 29.0 | 0.0 | 0.0 | 30 | 21 |
| 399 | 400 | Ningxia | Mainland China | 2020-01-30 21:30:00 | 21.0 | 0.0 | 1.0 | 30 | 21 |
| 400 | 401 | Inner Mongolia | Mainland China | 2020-01-30 21:30:00 | 20.0 | 0.0 | 0.0 | 30 | 21 |
| 401 | 402 | Xinjiang | Mainland China | 2020-01-30 21:30:00 | 17.0 | 0.0 | 0.0 | 30 | 21 |
| 402 | 403 | Guizhou | Mainland China | 2020-01-30 21:30:00 | 15.0 | 0.0 | 1.0 | 30 | 21 |
| 403 | 404 | Jilin | Mainland China | 2020-01-30 21:30:00 | 14.0 | 0.0 | 1.0 | 30 | 21 |
| 404 | 405 | Hong Kong | Hong Kong | 2020-01-30 21:30:00 | 12.0 | 0.0 | 0.0 | 30 | 21 |
| 405 | 406 | Taiwan | Taiwan | 2020-01-30 21:30:00 | 9.0 | 0.0 | 0.0 | 30 | 21 |
| 406 | 407 | Qinghai | Mainland China | 2020-01-30 21:30:00 | 8.0 | 0.0 | 0.0 | 30 | 21 |
| 407 | 408 | Macau | Macau | 2020-01-30 21:30:00 | 7.0 | 0.0 | 0.0 | 30 | 21 |
| 408 | 409 | Tibet | Mainland China | 2020-01-30 21:30:00 | 1.0 | 0.0 | 0.0 | 30 | 21 |
| 409 | 410 | Washington | US | 2020-01-30 21:30:00 | 1.0 | 0.0 | 0.0 | 30 | 21 |
| 410 | 411 | Illinois | US | 2020-01-30 21:30:00 | 2.0 | 0.0 | 0.0 | 30 | 21 |
| 411 | 412 | California | US | 2020-01-30 21:30:00 | 2.0 | 0.0 | 0.0 | 30 | 21 |
| 412 | 413 | Arizona | US | 2020-01-30 21:30:00 | 1.0 | 0.0 | 0.0 | 30 | 21 |
| 413 | 414 | NaN | Japan | 2020-01-30 21:30:00 | 11.0 | 0.0 | 1.0 | 30 | 21 |
| 414 | 415 | NaN | Thailand | 2020-01-30 21:30:00 | 14.0 | 0.0 | 5.0 | 30 | 21 |
| 415 | 416 | NaN | South Korea | 2020-01-30 21:30:00 | 6.0 | 0.0 | 0.0 | 30 | 21 |
| 416 | 417 | NaN | Singapore | 2020-01-30 21:30:00 | 10.0 | 0.0 | 0.0 | 30 | 21 |
| 417 | 418 | NaN | Vietnam | 2020-01-30 21:30:00 | 2.0 | 0.0 | 0.0 | 30 | 21 |
| 418 | 419 | NaN | France | 2020-01-30 21:30:00 | 5.0 | 0.0 | 0.0 | 30 | 21 |
| 419 | 420 | NaN | Nepal | 2020-01-30 21:30:00 | 1.0 | 0.0 | 0.0 | 30 | 21 |
| 420 | 421 | NaN | Malaysia | 2020-01-30 21:30:00 | 8.0 | 0.0 | 0.0 | 30 | 21 |
| 421 | 422 | Ontario | Canada | 2020-01-30 21:30:00 | 2.0 | 0.0 | 0.0 | 30 | 21 |
| 422 | 423 | British Columbia | Canada | 2020-01-30 21:30:00 | 1.0 | 0.0 | 0.0 | 30 | 21 |
| 423 | 424 | NaN | Cambodia | 2020-01-30 21:30:00 | 1.0 | 0.0 | 0.0 | 30 | 21 |
| 424 | 425 | NaN | Sri Lanka | 2020-01-30 21:30:00 | 1.0 | 0.0 | 0.0 | 30 | 21 |
| 425 | 426 | New South Wales | Australia | 2020-01-30 21:30:00 | 4.0 | 0.0 | 2.0 | 30 | 21 |
| 426 | 427 | Victoria | Australia | 2020-01-30 21:30:00 | 2.0 | 0.0 | 0.0 | 30 | 21 |
| 427 | 428 | Queensland | Australia | 2020-01-30 21:30:00 | 3.0 | 0.0 | 0.0 | 30 | 21 |
| 428 | 429 | Bavaria | Germany | 2020-01-30 21:30:00 | 4.0 | 0.0 | 0.0 | 30 | 21 |
| 429 | 430 | NaN | Finland | 2020-01-30 21:30:00 | 1.0 | 0.0 | 0.0 | 30 | 21 |
| 430 | 431 | NaN | United Arab Emirates | 2020-01-30 21:30:00 | 4.0 | 0.0 | 0.0 | 30 | 21 |
| 431 | 432 | NaN | Philippines | 2020-01-30 21:30:00 | 1.0 | 0.0 | 0.0 | 30 | 21 |
| 432 | 433 | NaN | India | 2020-01-30 21:30:00 | 1.0 | 0.0 | 0.0 | 30 | 21 |
| 433 | 434 | NaN | Italy | 2020-01-30 21:30:00 | 2.0 | 0.0 | 0.0 | 30 | 21 |
data[data['Day'] == 30].sum()
Sno 23895
Country Mainland ChinaMainland ChinaMainland ChinaMain...
Confirmed 9776
Deaths 213
Recovered 187
Day 1770
Hour 1239
dtype: object
Мы можем видеть, что число подтвержденных случаев для провинции Хубэй в Китае — 5806 на 30-е число. Количества смертей, выздоровевших и пострадавших, соответствует официальным на 30 января. Это означает, что в Confirmed уже включены люди, затронуте в предыдущие даты.
Создаем датасета с данными только за 30 января.
latest_data = data[data['Day'] == 30]
latest_data.head()
| Sno | Province/State | Country | Last Update | Confirmed | Deaths | Recovered | Day | Hour | |
|---|---|---|---|---|---|---|---|---|---|
| 375 | 376 | Hubei | Mainland China | 2020-01-30 21:30:00 | 5806.0 | 204.0 | 116.0 | 30 | 21 |
| 376 | 377 | Zhejiang | Mainland China | 2020-01-30 21:30:00 | 537.0 | 0.0 | 9.0 | 30 | 21 |
| 377 | 378 | Guangdong | Mainland China | 2020-01-30 21:30:00 | 393.0 | 0.0 | 11.0 | 30 | 21 |
| 378 | 379 | Henan | Mainland China | 2020-01-30 21:30:00 | 352.0 | 2.0 | 3.0 | 30 | 21 |
| 379 | 380 | Hunan | Mainland China | 2020-01-30 21:30:00 | 332.0 | 0.0 | 2.0 | 30 | 21 |
print('Подтвержденные случаи (весь мир): ', latest_data['Confirmed'].sum())
print('Смерти (весь мир): ', latest_data['Deaths'].sum())
print('Выздоровления (весь мир): ', latest_data['Recovered'].sum())
Подтвержденные случаи (весь мир): 9776.0
Смерти (весь мир): 213.0
Выздоровления (весь мир): 187.0
Данные датасета соответствуют официальным данным.
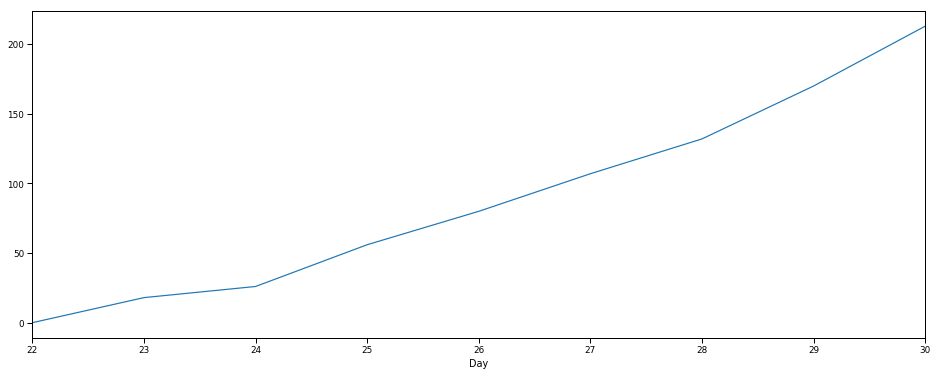
Посмотрим как коронавирус распространялся с течением времени.
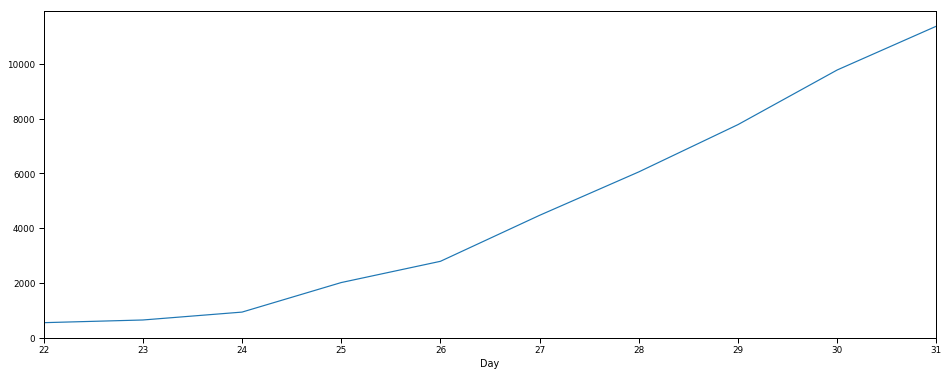
plt.figure(figsize=(16,6))
data.groupby('Day').sum()['Confirmed'].plot();
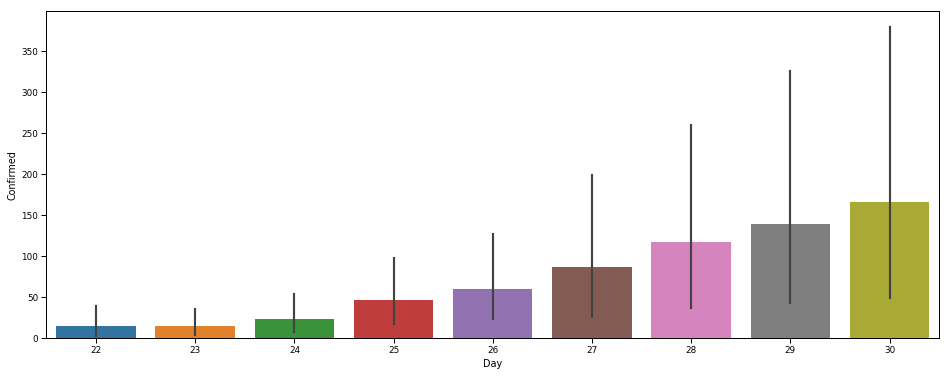
Со временем наблюдается экспоненциальный рост числа жертв короновируса.
plt.figure(figsize=(16,6))
sns.barplot(x='Day',y='Confirmed',data=data);
Глубокий разведочный анализ данных (EDA)
latest_data.groupby('Country').sum()
| Sno | Confirmed | Deaths | Recovered | Day | Hour | |
|---|---|---|---|---|---|---|
| Country | ||||||
| Australia | 1281 | 9.0 | 0.0 | 2.0 | 90 | 63 |
| Cambodia | 424 | 1.0 | 0.0 | 0.0 | 30 | 21 |
| Canada | 845 | 3.0 | 0.0 | 0.0 | 60 | 42 |
| Finland | 430 | 1.0 | 0.0 | 0.0 | 30 | 21 |
| France | 419 | 5.0 | 0.0 | 0.0 | 30 | 21 |
| Germany | 429 | 4.0 | 0.0 | 0.0 | 30 | 21 |
| Hong Kong | 405 | 12.0 | 0.0 | 0.0 | 30 | 21 |
| India | 433 | 1.0 | 0.0 | 0.0 | 30 | 21 |
| Italy | 434 | 2.0 | 0.0 | 0.0 | 30 | 21 |
| Japan | 414 | 11.0 | 0.0 | 1.0 | 30 | 21 |
| Macau | 408 | 7.0 | 0.0 | 0.0 | 30 | 21 |
| Mainland China | 12126 | 9658.0 | 213.0 | 179.0 | 930 | 651 |
| Malaysia | 421 | 8.0 | 0.0 | 0.0 | 30 | 21 |
| Nepal | 420 | 1.0 | 0.0 | 0.0 | 30 | 21 |
| Philippines | 432 | 1.0 | 0.0 | 0.0 | 30 | 21 |
| Singapore | 417 | 10.0 | 0.0 | 0.0 | 30 | 21 |
| South Korea | 416 | 6.0 | 0.0 | 0.0 | 30 | 21 |
| Sri Lanka | 425 | 1.0 | 0.0 | 0.0 | 30 | 21 |
| Taiwan | 406 | 9.0 | 0.0 | 0.0 | 30 | 21 |
| Thailand | 415 | 14.0 | 0.0 | 5.0 | 30 | 21 |
| US | 1646 | 6.0 | 0.0 | 0.0 | 120 | 84 |
| United Arab Emirates | 431 | 4.0 | 0.0 | 0.0 | 30 | 21 |
| Vietnam | 418 | 2.0 | 0.0 | 0.0 | 30 | 21 |
- Материковый Китай имеет ненулевые значения выздоровлений и смертей, которые можно изучить позже, создав отдельный набор данных
Провинции и регионы в которых нет зарегистрированных случаев заболевания.
data[data['Confirmed']==0]
| Sno | Province/State | Country | Last Update | Confirmed | Deaths | Recovered | Day | Hour | |
|---|---|---|---|---|---|---|---|---|---|
| 4 | 5 | Gansu | China | 2020-01-22 12:00:00 | 0.0 | 0.0 | 0.0 | 22 | 12 |
| 10 | 11 | Heilongjiang | China | 2020-01-22 12:00:00 | 0.0 | 0.0 | 0.0 | 22 | 12 |
| 12 | 13 | Hong Kong | China | 2020-01-22 12:00:00 | 0.0 | 0.0 | 0.0 | 22 | 12 |
| 15 | 16 | Inner Mongolia | China | 2020-01-22 12:00:00 | 0.0 | 0.0 | 0.0 | 22 | 12 |
| 18 | 19 | Jilin | China | 2020-01-22 12:00:00 | 0.0 | 0.0 | 0.0 | 22 | 12 |
| 22 | 23 | Qinghai | China | 2020-01-22 12:00:00 | 0.0 | 0.0 | 0.0 | 22 | 12 |
| 23 | 24 | Shaanxi | China | 2020-01-22 12:00:00 | 0.0 | 0.0 | 0.0 | 22 | 12 |
| 30 | 31 | Tibet | China | 2020-01-22 12:00:00 | 0.0 | 0.0 | 0.0 | 22 | 12 |
| 32 | 33 | Xinjiang | China | 2020-01-22 12:00:00 | 0.0 | 0.0 | 0.0 | 22 | 12 |
| 53 | 54 | Inner Mongolia | Mainland China | 2020-01-23 12:00:00 | 0.0 | 0.0 | 0.0 | 23 | 12 |
| 60 | 61 | Qinghai | Mainland China | 2020-01-23 12:00:00 | 0.0 | 0.0 | 0.0 | 23 | 12 |
| 68 | 69 | Tibet | Mainland China | 2020-01-23 12:00:00 | 0.0 | 0.0 | 0.0 | 23 | 12 |
| 77 | 78 | NaN | Philippines | 2020-01-23 12:00:00 | 0.0 | 0.0 | 0.0 | 23 | 12 |
| 78 | 79 | NaN | Malaysia | 2020-01-23 12:00:00 | 0.0 | 0.0 | 0.0 | 23 | 12 |
| 80 | 81 | NaN | Australia | 2020-01-23 12:00:00 | 0.0 | 0.0 | 0.0 | 23 | 12 |
| 81 | 82 | NaN | Mexico | 2020-01-23 12:00:00 | 0.0 | 0.0 | 0.0 | 23 | 12 |
| 82 | 83 | NaN | Brazil | 2020-01-23 12:00:00 | 0.0 | 0.0 | 0.0 | 23 | 12 |
| 115 | 116 | Qinghai | Mainland China | 2020-01-24 12:00:00 | 0.0 | 0.0 | 0.0 | 24 | 12 |
| 263 | 264 | NaN | Ivory Coast | 2020-01-27 20:30:00 | 0.0 | 0.0 | 0.0 | 27 | 20 |
- Интересно, что есть части материкового Китая, которые еще не были затронуты вирусом.
- Есть страны без подтвержденных случаев заражения, и мы отбросим их.
Провинции и регионы в которых есть минимум 1 зарегистрированный случай заболевания.
data = data[data['Confirmed'] != 0]
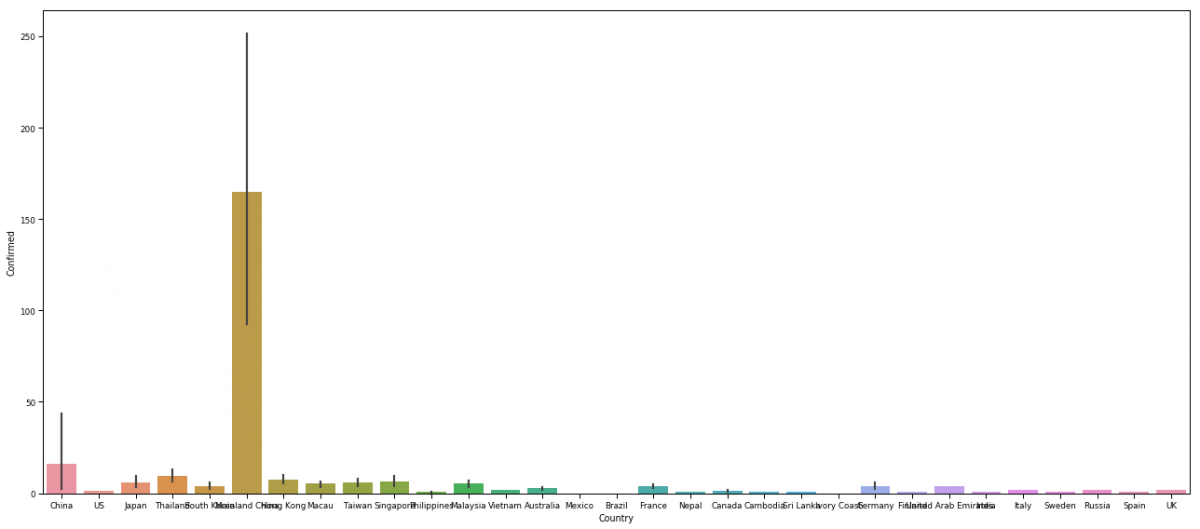
Количество зараженных в разных странах.
plt.figure(figsize=(18,8))
sns.barplot(x='Country',y='Confirmed',data=data)
plt.tight_layout()
- На графике показано то, что мы все знаем. Вирус больше всего затронул материковый Китай, однако есть сообщения о жертвах в соседних странах, что говорит о распространении вируса.
- Есть также случаи, подтвержденные в странах, которые далеко, таких как США, Таиланд, Япония и т. Д. Интересно, как вирус попал туда. Я предполагаю, что кто-то был в Ухане или близлежащем районе во время распространения вируса и увез его с собой домой, эта вспышка действительно опасна.
Количество зараженных в разных регионах.
import plotly.express as px
fig = px.bar(data, x='Province/State', y='Confirmed')
fig.show()
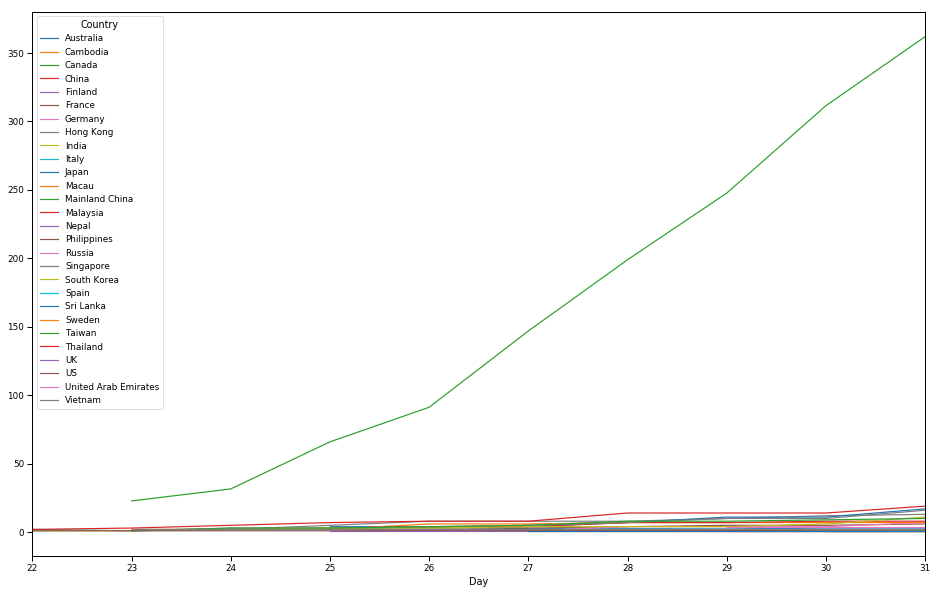
Анализ роста коронавируса в каждой стране
pivoted = pd.pivot_table(data, values='Confirmed', columns='Country', index='Day')
pivoted.plot(figsize=(16,10));
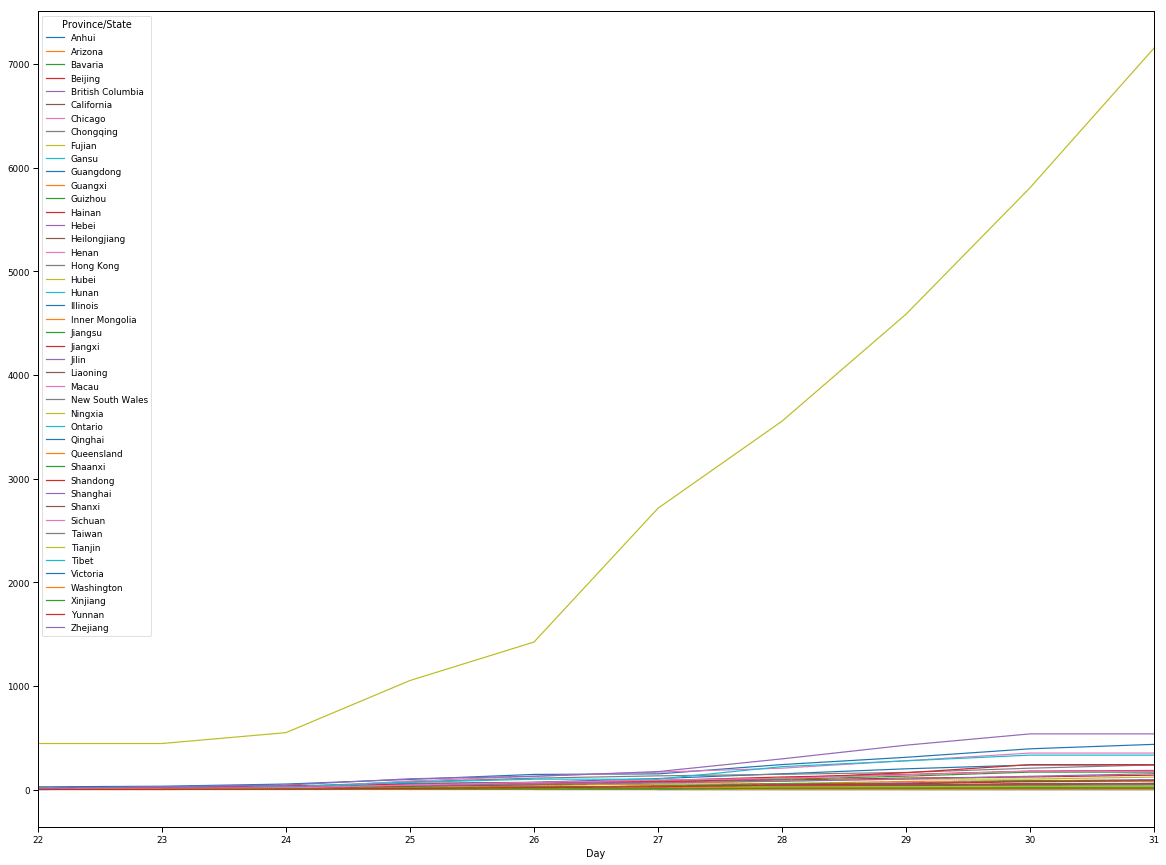
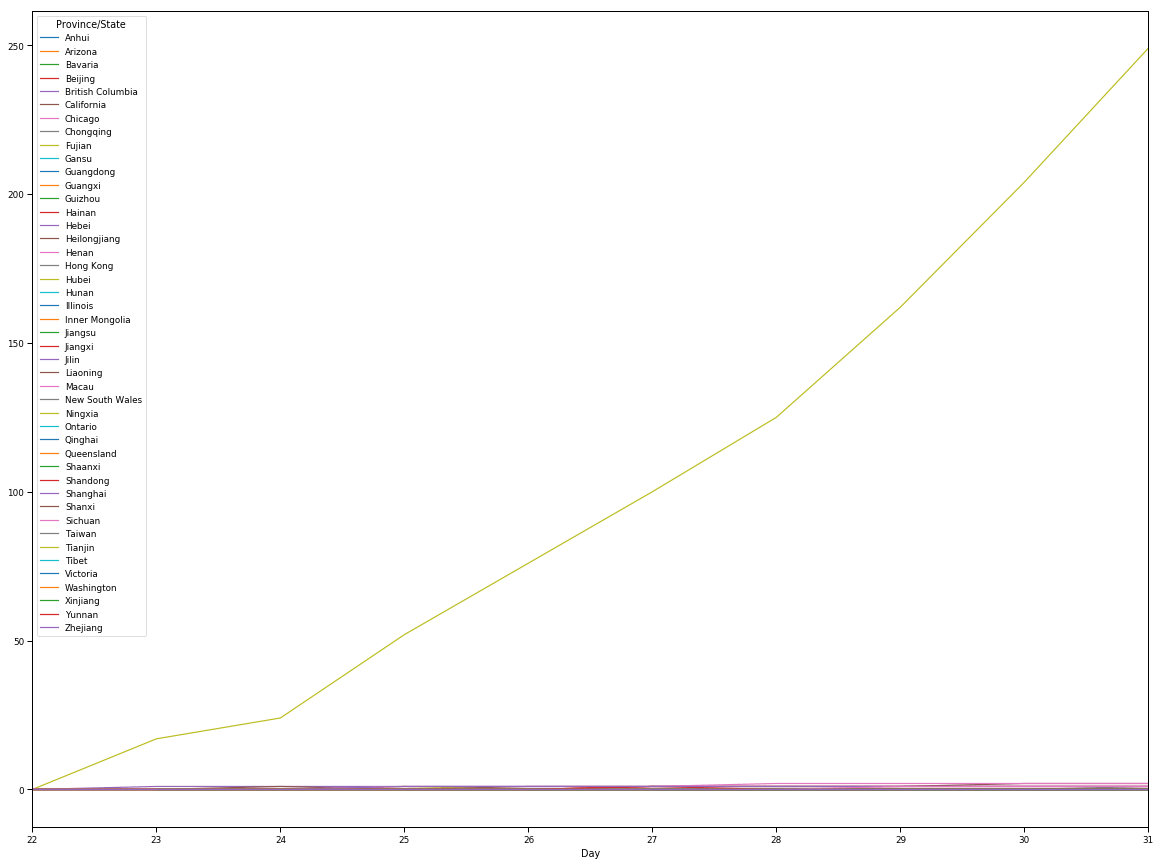
Визуализация вспышки в провинциях/регионах
pivoted = pd.pivot_table(data, values='Confirmed', columns='Province/State', index='Day')
pivoted.plot(figsize=(20,15));
- Hubei, наиболее пострадавшая провинция.
- Также в подтвержденных случаях наблюдается тенденция к росту, и кажется, что состояние ухудшается.
Теперь давайте посмотрим на страны, которые были затронуты изначально, и страны, в которые сейчас проник коронный вирус.
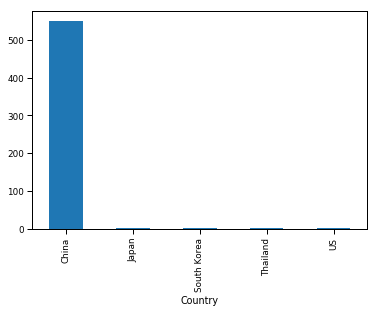
data[data['Day'] == 22]['Country'].unique()
array(['China', 'US', 'Japan', 'Thailand', 'South Korea'], dtype=object)
Итак, в первый день, 22 января заражения были обнаружены в Китае, США, Японии, Таиланде
temp = data[data['Day'] == 22]
temp.groupby('Country').sum()['Confirmed'].plot.bar()
Посмотрим на последние данные.
data[data['Day'] == 30]['Country'].unique()
array(['Mainland China', 'Hong Kong', 'Taiwan', 'Macau', 'US', 'Japan',
'Thailand', 'South Korea', 'Singapore', 'Vietnam', 'France',
'Nepal', 'Malaysia', 'Canada', 'Cambodia', 'Sri Lanka',
'Australia', 'Germany', 'Finland', 'United Arab Emirates',
'Philippines', 'India', 'Italy'], dtype=object)
Здесь мы видим, что вспышка распространилась в 23 странах к 30 января.
Рассмотрим только материковый Китай
data_main_china = latest_data[latest_data['Country']=='Mainland China']
Рассчитаем процент смертей.
(data_main_china['Deaths'].sum() / data_main_china['Confirmed'].sum())*100
2.205425553944916
Теперь процент выздоровлений.
(data_main_china['Recovered'].sum() / data_main_china['Confirmed'].sum())*100
1.8533857941602818
- Мы можем видеть, что процент смертности от коронавируса — 2%, поэтому он не такой смертоносный, как другие вирусные вспышки.
- Поскольку зарегистрировано мало случаев излечения, процент выздоровления составляет 1,87, это страшно. Хотя цифра может сильно вырасти, ведь 96% сейчас не попадают ни в одну из групп.
Где произошло большинство смертей
data_main_china.groupby('Province/State')['Deaths'].sum().reset_index(
).sort_values(by=['Deaths'],ascending=False).head()
| Province/State | Deaths | |
|---|---|---|
| 12 | Hubei | 204.0 |
| 10 | Heilongjiang | 2.0 |
| 11 | Henan | 2.0 |
| 9 | Hebei | 1.0 |
| 1 | Beijing | 1.0 |
Количество смертей по дням.
plt.figure(figsize=(16,6))
data.groupby('Day').sum()['Deaths'].plot();
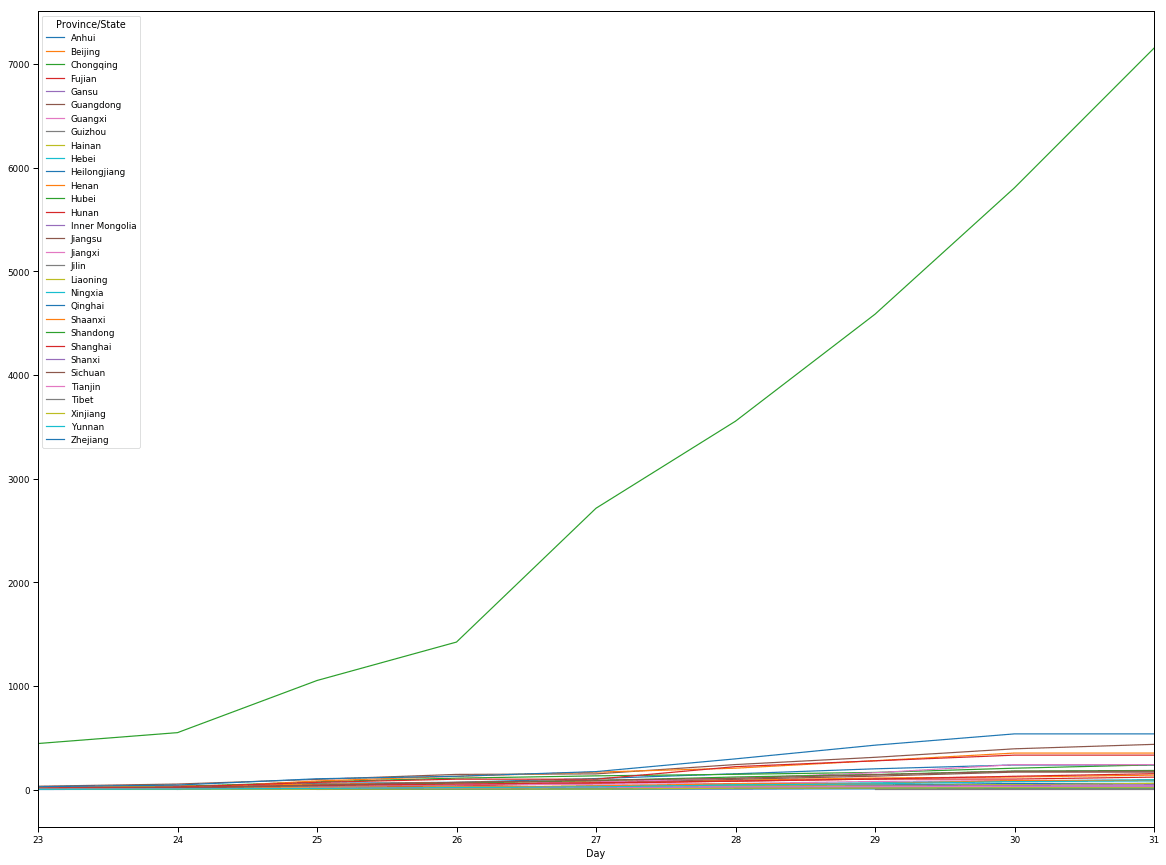
График заболеваний в материковом Китае.
pivoted = pd.pivot_table(data[data['Country']=='Mainland China'] , values='Confirmed', columns='Province/State', index='Day')
pivoted.plot(figsize=(20,15))
pivoted = pd.pivot_table(data, values='Deaths', columns='Province/State', index='Day')
pivoted.plot(figsize=(20,15));
Что дальше
Скачайте coronavirus.ipynb и данные по ссылке в начале статьи. Попробуйте построить свои графики и таблицы.